La bellezza matematica dei polimini risiede nella loro capacità di generare complessità infinita da regole semplici, offrendo un microcosmo dove esplorare i principi fondamentali della forma e della struttura. La classificazione morfologica dei polimini rappresenta un ponte tra matematica pura e applicata. L'analisi di indicatori quantitativi e qualitativi consente di categorizzare queste strutture discrete secondo caratteristiche geometriche fondamentali.
Il rapporto tra perimetro e area di un polimino, il suo numero di elementi $n$, è un indicatore semplice ma informativo per una classificazione morfologica: misura la compattezza della forma, tra un valore minimo $\frac{4\sqrt n}{n}$ per le forme quadrate a $\frac{2n+2}{n}$ per le forme a asta, assumendo valori massimi per forme a "T", "X" o strutture dendritiche.
Altri semplici indicatori sono:
- il rapporto tra altezza massima e larghezza massima (aspect ratio) per l'allungamento direzionale;
def aspect_ratio(S): lst_x, lst_y = zip(*S) width = max(lst_x) - min(lst_x) + 1 height = max(lst_y) - min(lst_y) + 1 return height/width
- il rapporto tra l'area del polimino e quella del più piccolo rettangolo che lo contiene;
def A_R(S): lst_x, lst_y = zip(*S) width = max(lst_x) - min(lst_x) + 1 height = max(lst_y) - min(lst_y) + 1 return len(S)/(width*height)
- il rapporto tra l'area del polimino e la lunghezza della diagonale del più piccolo rettangolo che lo contiene;
def A_Rdiag(S): lst_x, lst_y = zip(*S) width = max(lst_x) - min(lst_x) + 1 height = max(lst_y) - min(lst_y) + 1 return len(S)/np.sqrt(width**2 + height**2)
- l'indice di ramificazione, ovvero il rapporto tra il numero di vertici grado 3 o 4 e il numero totale di vertici;
def vertici_molt(S): count = dict() for (x, y) in S: for v in {(x, y), (x+1, y), (x, y+1), (x+1, y+1)}: count[v] = 1 if v not in count else count[v]+1 return count def n_vertici_molt(S,k): count = vertici_molt(S) return len({v for v, c in count.items() if c == k})
- il rapporto tra l'area del bordo esterno, o del bordo interno o dell'interno, e quella del polimino oppure il rapporto tra l'area del bordo esterno e quella del bordo interno.
Un indicatore più sofisticato, è il raggio di girazione definito come: \[ R_g = \sqrt{\frac{1}{n} \sum_{i=1}^n \|\mathbf{r}_i - \mathbf{r}_0\|^2} \] dove $\mathbf{r}_0$ è il centro di massa.
def R_g(S):
if not S:
return 0.0
r = np.array(list(S))
r_0 = np.mean(r, axis=0)
# Distanza quadratica media dal centro
squared_distances = np.sum((r - r_0) ** 2, axis=1)
return np.sqrt(np.mean(squared_distances))
n = 30
S, _ = genera(n)
plot_S(S)
print(perim(S)/n, R_g(S))

1.2666666666666666 2.6627053911388696
Anche il raggio di girazione, una misura che cattura globalmente la "forma" del polimino, da valori bassi per quelli più compatti, cresce per polimini sempre più allungati.
I precedenti indicatori possono essere valutati in una pagina dinamica interattiva insieme ad altri più complessi come i seguenti:
- i momenti di inerzia che misurano come la "massa", cioè le celle, è distribuita rispetto agli assi;
def Inerzia(S): r = np.array(list(S)) x_CM, y_CM = np.mean(r, axis=0) # centro di massa I_xx = np.sum((r[:, 0] - y_CM)**2) # Rispetto asse x I_yy = np.sum((r[:, 1] - x_CM)**2) # Rispetto asse y I_xy = -np.sum((r[:, 0] - x_CM) * (r[:, 1] - y_CM)) # Prodotto di inerzia return I_xx, I_yy, I_xy
- l'anisotropia, il grado di direzionalità/allungamento, se minore di 0.1 indica forma circolare, maggiore di 1 indica forma allungata;
def anisotropia(S): I_xx, I_yy, _ = Inerzia(S) # Anisotropia (0 = isotropo, 1 = massima anisotropia) return abs(I_xx - I_yy) / (I_xx + I_yy) if (I_xx + I_yy) > 0 else 0
- la direzione di massima estensione e la direzione di minima estensione, ortogonali tra loro;
def direz(S): I_xx, I_yy, I_xy = Inerzia(S) I = np.array([ [I_xx, I_xy], [I_xy, I_yy] ]) eigenvalues, eigenvectors = np.linalg.eigh(I) return { 'Imin': eigenvalues[0], # Momento principale minore 'Imax': eigenvalues[1], # Momento principale maggiore 'Imax/Imin': eigenvalues[1]/eigenvalues[0], # Rapporto di allungamento 'axis1': eigenvectors[:, 0], # Direzione primo asse principale 'axis2': eigenvectors[:, 1] # Direzione secondo asse principale }
- il grado di simmetria rispetto a una cella o rispetto a linee di celle parallele agli assi coordinati;
def gradoSimmC(S,C): lstx, lsty = zip(*S) x_C, y_C = C lstx, lsty = [2*x_C-x for x in lstx], [2*y_C-y for y in lsty] S1 = set(zip(lstx, lsty)) return len(S & S1)/len(S) def gradoSimmAss(S,asse,C): lstx, lsty = zip(*S) x_C, y_C = C if asse == 'y': lstx = [2*x_C-x for x in lstx] else: lsty = [2*y_C-y for y in lsty] S1 = set(zip(lstx, lsty)) return len(S & S1)/len(S)
- il numero di concavità, ovvero il numero di "rientranze" nel contorno; si potrebbe determinare come il numero di componenti connesse del complementare del polimino rispetto al rettangolo che ne è l'involucro;
def Cbox(S): lst_x,lst_y = zip(*S) xM, xm = max(lst_x), min(lst_x) yM, ym = max(lst_y), min(lst_y) return {(x, y) for x in range(xm, xM + 1) for y in range(ym, yM + 1) if (x,y) not in S} def connected_components(S): visited = set() components = [] def dfs(s, comp): stack = [s] while stack: x, y = stack.pop() if (x, y) in visited: continue visited.add((x, y)) comp.add((x, y)) for (x,y) in [(x+1,y), (x-1,y), (x,y+1), (x,y-1)]: if (x,y) in S and (x,y) not in visited: stack.append((x,y)) for s in S: if s not in visited: comp = set() dfs(s, comp) components.append(comp) return components len([Si for Si in connected_components(Cbox(S))])
- il numero di "buchi"; ad esempio considerando quelle componenti connesse del complementare del polimino rispetto al polimino che ne è l'involucro convesso, o anche del rettangolo minimo che lo contiene, il cui bordo esterno è parte del polimino, con ciò considerando come buchi anche le "insenature" o rientranze inaccessibili;
len([Si for Si in connected_components(invConv(S)-S) if bordoEst(Si).issubset(S)])
- la massima distanza tra due celle del polimino considerando:
- la massima distanza euclidea;
def max_dist_euclid(S): S = list(S) maxd = 0 coppia = None for i in range(len(S)): x1, y1 = S[i] for j in range(i+1, len(S)): x2, y2 = S[j] d = ((x1 - x2)**2 + (y1 - y2)**2)**0.5 if d > maxd: maxd = d coppia = (S[i], S[j]) return maxd, coppia
- la massima distanza rispetto a $d((x_1,y_1),(x_2,y_2))=|x_2-x_1|+|y_2-y_1|$;
def max_dist_manhattan(S): S = list(S) maxd = 0 coppia = None for i in range(len(S)): x1, y1 = S[i] for j in range(i+1, len(cells)): x2, y2 = S[j] d = abs(x1 - x2) + abs(y1 - y2) if d > maxd: maxd = d coppia = (S[i], S[j]) return maxd, coppia
- la distanza massima in “passi ortogonali” (come BFS all’interno del polimino), vero diametro topologico del polimino.
def max_geodesic_distance(S): # BFS per trovare il punto più distante da un qualunque punto def bfs(start): visited = {start: 0} queue = deque([start]) farthest = (start, 0) while queue: xc,yc = queue.popleft() for (x,y) in {(xc+1,yc),(xc,yc+1),(xc-1,yc),(xc,yc-1)} & S: if (x,y) not in visited: visited[(x,y)] = visited[(xc,yc)] + 1 queue.append((x,y)) if visited[(x,y)] > farthest[1]: farthest = ((x,y), visited[(x,y)]) return farthest # Step 1: prendi una cella a caso start = next(iter(S)) a, _ = bfs(start) # Step 2: BFS da quella cella per trovare la distanza massima effettiva b, dist = bfs(a) return dist, (a, b)
- la massima distanza euclidea;
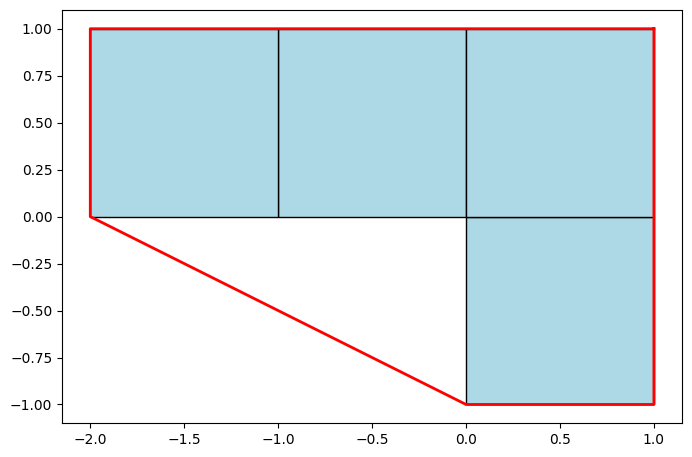
Infine possiamo considerare il rapporto tra area del polimino e area del suo involucro convesso che indica la convessità del polimino. Il metodo .ConvexHull() della libreria scipy.spatial fornisce le coordinate del poligono involucro (hull) convesso di un insieme di punti.
def Hull_conv(S):
# --- 1. Estrarre tutti i vertici unici delle celle ---
V = set()
for x, y in S:
V |= {(x, y),(x + 1, y),(x, y + 1),(x + 1, y + 1)}
P = np.array(list(V))
hull = ConvexHull(P)
return P[hull.vertices]
n = 4
S, _ = genera(n)
hull_points = Hull_conv(S)
N = len(hull_points)
hull_area = 0.5 * abs(
sum(hull_points[i][0] * hull_points[(i + 1) % N][1] for i in range(N)) -
sum(hull_points[i][1] * hull_points[(i + 1) % N][0] for i in range(N))
)
print(n/hull_area)
fig, ax = plt.subplots(figsize=(8, 8))
ax.set_aspect('equal')
for (x, y) in S:
ax.add_patch(plt.Rectangle((x, y), 1, 1,
facecolor='lightblue',
edgecolor='black'))
ax.plot(np.append(hull_points[:, 0], hull_points[0, 0]),
np.append(hull_points[:, 1], hull_points[0, 1]), 'r-', linewidth=2)
plt.show()
0.8
- POLYOMINOES, by Gill Barequet, Solomon W. Golomb, and David A. Klarner